研究背景:花生作为一种重要的农作物,其产量的提升对于全球食品供应具有重要意义。HPW、HSW和SP是影响花生产量的三个主要因素。
实验材料:
-
使用了重组近交系(RILs)群体,这些RILs源自“Silihong”(A. hypogaea var. fastigiate)和“Jinonghei 3”(A. hypogaea var. hypogaea)的杂交。
-
利用美国小核心种质集合作为关联分析的群体。
实验方案:
-
高密度遗传连锁图谱构建:通过重新测序RILs,构建了一个包含4499个箱(bins)的高密度遗传连锁图谱,覆盖20条染色体,总长度为1712.32 cM。
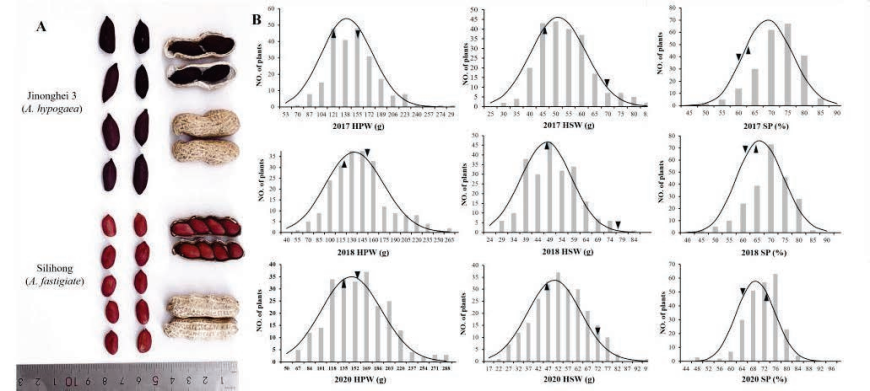
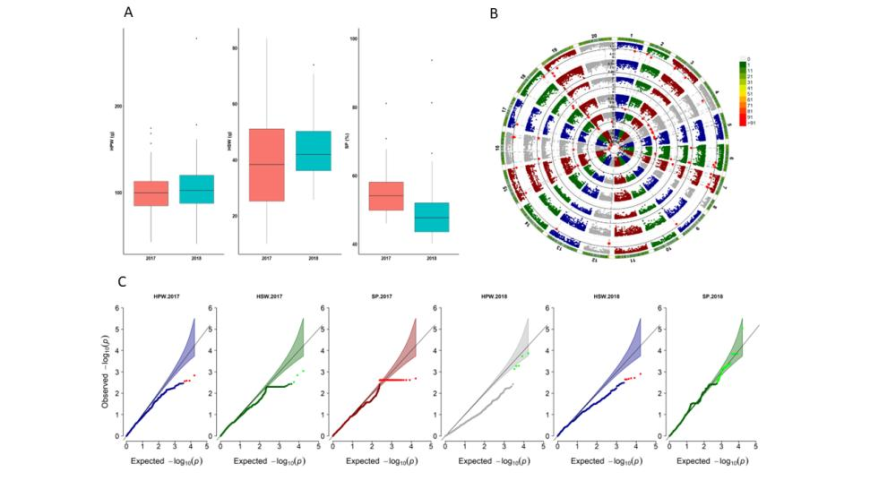
2. 数量性状位点(QTL)分析:在三个不同的环境下,共鉴定出46个与HPW、HSW和SP相关的QTLs。其中,一些主要QTLs如qHPW5.2、qSP7.1和qSP18.2等,显示出较高的表型变异解释率(PVE)。

3. 全基因组关联研究(GWAS):使用美国小核心种质集合进行GWAS,以验证QTL定位的准确性,并在两个环境中鉴定出与HPW、HSW和SP显著相关的115个单核苷酸多态性(SNPs)。
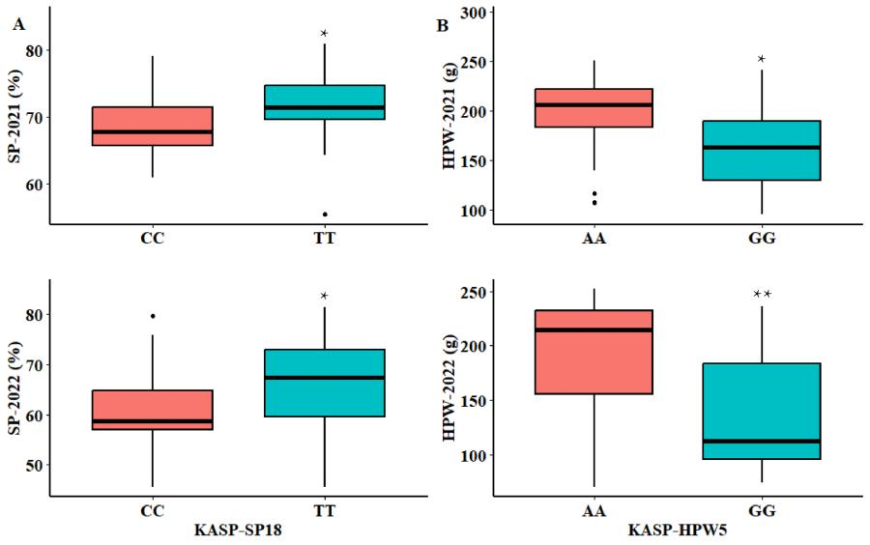
4. KASP标记开发与验证:基于QTL区域,开发了三个KASP标记,并在花生品种和地方品种中进行了验证,这些标记与HPW、HSW和SP有显著关联。
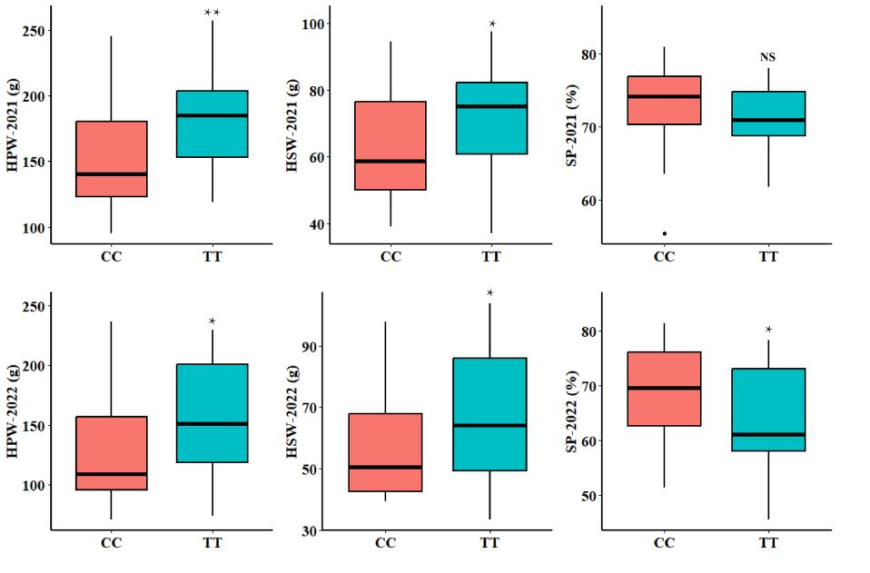
上图展示了KASP-SP7标记对花生小组中SP、HPW和HSW的影响。包括统计图表显示KASP-SP7不同等位基因对目标性状的影响,并用星号表示显著性水平。
上图展示了KASP-SP18和KASP-HPW5标记在花生小组中的验证效果。包括两个子图,A展示KASP-SP18对SP的影响,B展示KASP-HPW5对HPW的影响,同样使用统计图表和显著性标记。
研究结果:
-
确定了多个与HPW、HSW和SP相关的QTLs和SNPs,其中一些是首次发现。
-
通过GWAS验证了QTL定位的准确性,并且发现了一些QTLs和SNPs的共定位。
-
开发的KASP标记为花生分子辅助育种提供了有用的工具。
结论:本研究不仅为理解花生HPW、HSW和SP的遗传基础提供了新的见解,而且开发的KASP标记有助于加速花生的分子辅助育种进程。
随着分子生物学技术的不断进步,KASP技术将在现代农业发展中扮演越来越重要的角色。东盛生物致力于推动生物科技在农业领域的应用,KASP试剂的推出是我们对创新和精准农业的承诺。我们期待与广大科研工作者和育种专家携手合作,共同开启农业科技的新篇章。